Os Coronavírus
Estrutura Viral, Classificação e Origem
Christiane Lourenço Nogueira (Theraphage Inc. / University of Waterloo)
27/05/2020
Os coronavírus são vírus pleomórficos e envelopados, sendo seu material genético constituído por uma fita simples de RNA com 26 a 32 kilobases, possuindo, desta forma, o maior genoma dentre os vírus de RNA. O seu genoma codifica quatro principais proteínas estruturais: Spike (S), Membrana (M), Envelope (E) e Nucleocapsídeo (N) (Figura 1)\(^{1,2}\).
As proteínas Spike possuem forma de espículas, as quais saem do envelope viral, conferindo ao vírus um formato de coroa solar, por isso a denominação coronavírus\(^{3}\). A proteína Spike é fundamental no processo de infecção. De forma geral, esta proteína liga-se a receptores celulares nas células dos hospedeiros, permitindo a entrada do vírus nestas células. Uma vez dentro das células, os vírus replicam-se e geram novas partículas virais, as quais vão infectar outras células do hospedeiro, e assim sucessivamente. Além disso, a proteína Spike é o principal componente antigênico capaz de induzir respostas imunes no hospedeiro. Desta forma, esta proteína é fundamental tanto no processo de infecção viral quanto na patogênese, sendo, por esta razão, um importante alvo para tratamentos e vacinas contra os coronavírus.
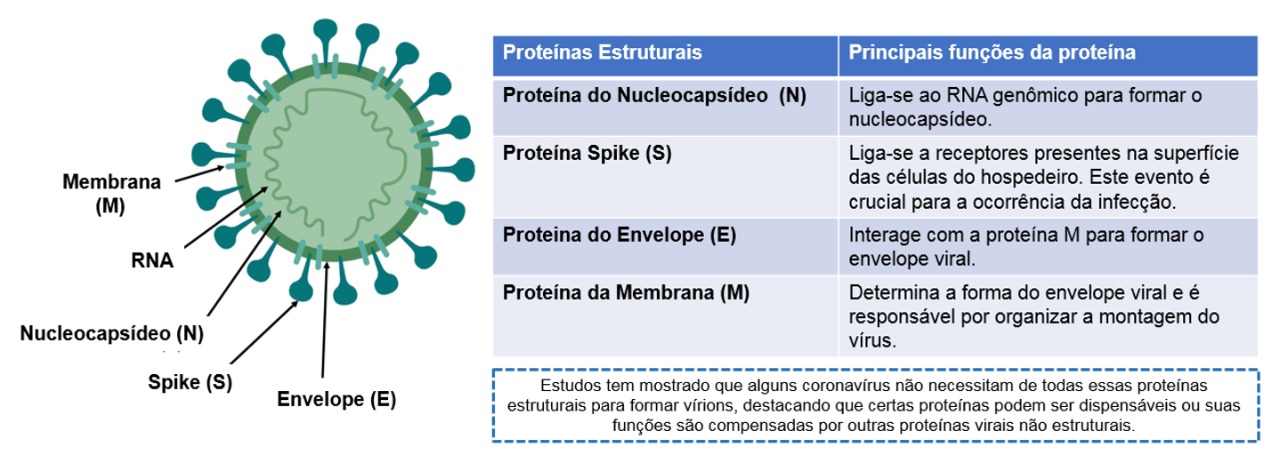
Figura 1. Estrutura geral dos coronavírus (adaptado de Seah et al., 2020\(^{4}\)).
Os coronavírus pertencem ao Reino Riboviria, à ordem Nidovirales e à família Coronaviridae, a qual é constituída de duas subfamílias, a Orthocoronavirinae e a Letovirinae. Esta última é subdividida em diferentes gêneros, incluindo Alpha-, Beta-, Gamma- e Delta-coronavírus. O gênero Beta-coronavírus, por sua vez, é dividido em quatro linhagens (A, B, C and D)\(^{5}\).
De modo geral, os Gamma- e Delta-coronavírus infectam aves e roedores, enquanto a maioria dos Alfa- e Beta-coronavírus infectam diferentes espécies de mamíferos, incluindo morcegos, gatos, cachorros, porcos, cavalos, baleias e o homem\(^{6,7}\). A grande maioria destes hospedeiros pode desenvolver doenças respiratórias, entéricas, hepáticas ou neurológicas, com diferentes graus de severidade\(^{3}\). Em alguns mamíferos e aves, os coronavírus causam doenças letais com alto impacto na indústria agropecuária, incluindo o vírus da bronquite infecciosa aviária (IBV – do inglês Infectious Bronchitis Virus), vírus da gastroenterite transmissível (TGEV – Transmissible Gastroenteritis Virus), vírus da diarréia epidêmica suína (PEDV – Porcine Epidemic Diarrhea Virus) e o vírus da síndrome da diarréia aguda suína (SADS-CoV – Swine Acute Diarrhea Syndrome Coronavirus)\(^{8,9,10}\). Estima-se que o PEDV levou à redução de 3,21% no plantel suíno nos EUA entre setembro de 2012 e agosto de 2014, enquanto o IBV causou uma perda de cerca de 1.000 dólares a cada mil aves em fazendas no Brasil\(^{11,12}\). No homem, os coronavírus causam principalmente doenças no trato respiratório superior, com sintomas semelhantes a um resfriado comum, e no trato respiratório inferior, levando a quadros de bronquite, pneumonia e síndrome respiratória aguda grave\(^{13}\). Os sete coronavírus conhecidos até o momento como patogênicos ao homem, e as doenças que eles causam, estão descritos na Figura 2.
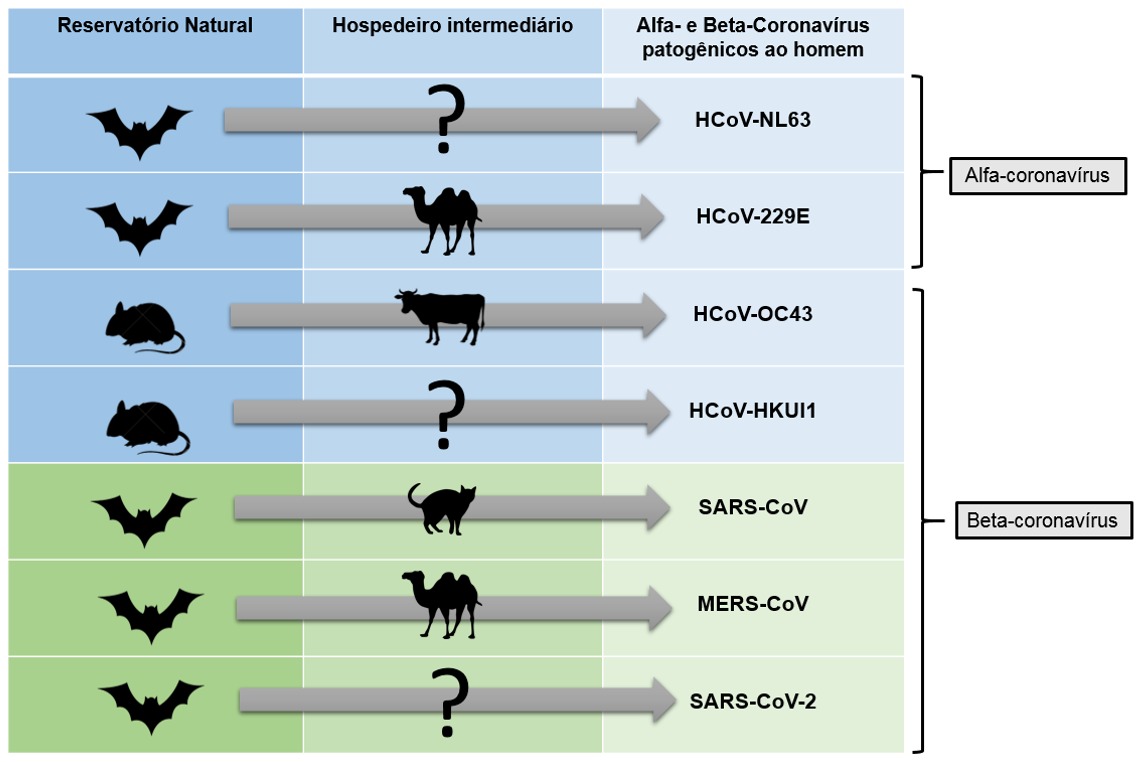
Figura 2. Coronavirus patogênicos ao homem e suas respectivas doenças.
Estudos filogenéticos têm indicado que a grande maioria dos Alfa- e Beta-coronavírus que causam doenças no homem apresentam os morcegos ou os roedores como ancestral comum, os quais são importantes reservatórios naturais desses vírus e não apresentam manifestação clínica de doença\(^{7,10,14,15}\). Alguns coronavírus cruzaram a barreira entre espécies animais-homem e progrediram para estabelecer doenças zoonóticas em humanos. Neste processo, outros animais podem atuar como hospedeiros intermediários e reservatórios de vírus\(^{3}\), como mostra a Figura 3.
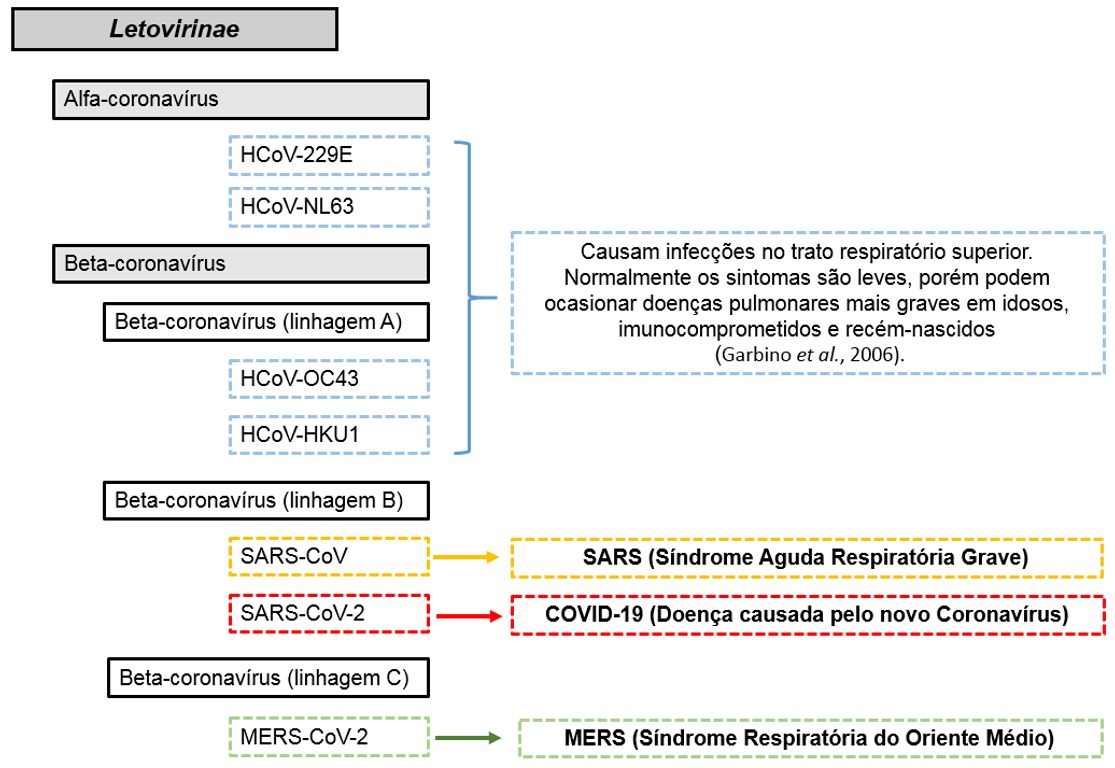
Figura 3. Prováveis reservatórios naturais e hospedeiros intermediários dos sete Alfa- e Beta-coronavírus conhecidos até o momento como patogênicos ao homem. Dromedários, animais bovinos e civetas são, provavelmente, hospedeiros intermediários de coronavírus (adapatado de Corman et al., 2018\(^{7}\)).
Com o advento da tecnologia de sequenciamento de próxima geração (nova abordagem de sequenciamento em larga escala) e o aumento da vigilância de espécies de animais selvagens, mais de 200 novos coronavírus foram identificados em morcegos, e aproximadamente 35% do viroma de morcego sequenciado até o momento é composto por coronavírus\(^{16}\). Além disso, morcegos constituem cerca de um quarto da diversidade de mamíferos e habitam todos os continentes, exceto o Antártico\(^{10}\). Considerando que as três pandemias (SARS, MERS e COVID-19) causadas por coronavírus apresentam o morcego como reservatório natural, e que neste animal já foram identificados diversos tipos de coronavírus, os morcegos representam uma importante fonte para que novos coronavírus possam causar futuras doenças infecciosas no homem.
Referências
[1] Schoeman, D., Fielding, B. C. (2019). Coronavirus envelope protein: current knowledge. Journal of Virology, 16:69.
[2] Wu, F. et al. (2020). Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome. Nature, 265-269.
[3] Malik, Y. A. (2020). Properties of Coronavirus and SARS-CoV-2. Malaysian Journal of Pathology, 42(1), 3-11.
[4] Seah, I., Su, X. & Lingam, G. (2020). Revisiting the dangers of the coronavirus in the ophthalmology practice.
[5] Gorbalenya et al. (2020). Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nature Microbiology, 5(4), 536‐544.
[6] Perlman, S., Netland, J. Coronaviruses post-SARS: update on replication and pathogenesis. (2009). Nature Reviews Microbiology, 7, 439-450.
[7] Corman, V. M., Muth, D., Niemeyer, D., & Drosten, C. (2018). Hosts and Sources of Endemic Human Coronaviruses. Advances in virus research, 100, 163–188.
[8] Pradesh, U., Upadhayay, P. D. D., Vigyan, P. C. (2014). Coronavirus infection in equines: A review. Asian Journal of Animal and Veterinary Advances, 9(3), 164-76.
[9] Lee C. (2015). Porcine epidemic diarrhea virus: An emerging and re-emerging epizootic swine virus. Virology Journal, 12(1), 193.
[10] Banerjee, A., Kulcsar, K., Misra, V., Frieman, M., & Mossman, K. (2019). Bats and Coronaviruses. Viruses, 11(1), 41.
[11] Schulz L.L., Tonsor G.T. (2015). Assessment of the economic impacts of porcine epidemic diarrhea virus in the United States. J. Anim. Sci., 93, 5111–5118.
[12] Colvero L.P., Villarreal L.Y.B., Torres C.A., Brandao P.E. (2015). Assessing the economic burden of avian infectious bronchitis on poultry farms in Brazil. Revue Scientifique et Technique de l’OIE, 34:993-999.
[13] Schoeman, D., Fielding, B. C. (2019). Coronavirus envelope protein: current knowledge. Journal of Virology.
[14] Calisher, C. H., Childs, J. E., Field, H. E., Holmes, K. V., and Schountz, T. (2006). Bats: important reservoir hosts of emerging viruses. Clinical Microbiology Reviews, 19, 531-545.
[15] Woo, P. C., Lau, S. K., Lam, C. S., Lau, C. C., Tsang, A. K., Lau, J. H., Bai, R., Teng, J. L., Tsang, C. C., Wang, M., Zheng, B. J., Chan, K. H., & Yuen, K. Y. (2012). Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus. Journal of virology, 86(7), 3995–4008.
[16] Chen, L., Liu, B., Yang, J., & Jin, Q. (2014). DBatVir: the database of bat-associated viruses. Database : the journal of biological databases and curation, 2014, bau021.